Paper
https://www.informingscience.org/Publications/5446
InformingSciJ - Healthcare Biclustering of Predictive Gene Expression Using LSTM Based Support Vector Machine
Healthcare Biclustering of Predictive Gene Expression Using LSTM Based Support Vector Machine Aim/PurposeThe major goal of this work is to establish prediction patterns that can influence better diagnosis and treatment strategies using unidentified interac
www.informingscience.org
논문 “Healthcare Biclustering of Predictive Gene Expression Using LSTM-SVM Hybrid”(2025)은 **LSTM(Long Short-Term Memory)**과 **SVM(Support Vector Machine)**을 결합한 하이브리드 모델을 사용하여 **유전자 발현 데이터의 바이클러스터링(biclustering)**을 수행하는 새로운 방법을 제안합니다. 이 모델은 특히 **시간 의존성(temporal dependencies)**과 패턴 인식 능력을 동시에 반영하여 예측 성능과 해석 가능성을 향상시키는 것을 목표로 합니다.
🔍 핵심 목표
- 유전자 간의 숨겨진 상호작용 및 발현 패턴을 발견하여 질병 진단과 치료 전략에 기여
- 정적인 분석법이 포착하지 못하는 시계열적 유전자 발현 패턴을 학습
- 기존의 HMM, RNN, SVM 등과 비교해 더 높은 정확도와 실루엣 점수(silhouette score) 달성
핵심 방법론: LSTM-SVM 하이브리드 바이클러스터링
< ALGORITHM: LSTM-SVM BICLUSTERING >
Obtain LSTM-generated embeddings representing temporal features of genes.
Utilize the LSTM embeddings to initialize an initial set of gene clusters.
For each initialized gene cluster:
Train the SVM on the gene expression profiles to optimize and refine the clusters.
Obtain refined SVM-generated clusters within the temporal context.
Iteratively refine the gene clusters by repeating
Reapply LSTM to update temporal features based on refined SVM clusters.
Refine clusters with SVM to enhance accuracy and precision
유전자의 시간적 특징을 나타내는 LSTM 생성 임베딩을 얻습니다
LSTM 임베딩을 활용하여 초기 유전자 클러스터 세트를 초기화하십시오
각 초기 유전자 클러스터에 대해 :
유전자 발현 프로파일에서 SVM을 훈련하여 클러스터를 최적화하고 개선하십시오.
시간적 컨텍스트 내에서 정제 된 SVM 생성 클러스터를 얻습니다.
반복하여 유전자 클러스터를 반복적으로 개선합니다.
정제 된 SVM 클러스터를 기반으로 시간적 기능을 업데이트하기 위해 LSTM을 다시 적용하십시오.
정확도와 정밀도를 향상시키기 위해 SVM으로 클러스터를 개선하십시오.1. LSTM을 통한 시계열 표현 학습
- 유전자 발현 데이터는 시간 축을 따라 정렬되어 LSTM의 입력으로 사용됨
- 반드시 진짜 시계열일 필요는 없고,
"임의로 시계열로 가정"하여 LSTM을 사용하는 것이 가능하며,
실제로 이 논문에서도 시간 순서가 보장되지 않은 sample 간 발현값에 "시계열적 가정"을 적용한 사례로 보입니다. - 예: 각 sample이 환자라면, 나이순/증상순 등 인위적 순서를 부여하여 입력 가능
- 반드시 진짜 시계열일 필요는 없고,
- 각 유전자에 대해 시계열적 특성을 반영한 **임베딩(embedding)**을 생성
- LSTM 구조:
- 입력 차원: 유전자 수
- 출력: 시간 종속 정보를 내포한 잠재 벡터
논문에서 제안한 LSTM-SVM 하이브리드 모델에서 LSTM(Long Short-Term Memory)은 유전자 발현 데이터를 처리하여 **시간적 패턴을 반영한 임베딩(embedding)**을 생성합니다. 여기서 임베딩은 각 유전자의 시계열 발현 패턴을 요약한 고차원 표현 벡터로, 이후 SVM에서 클러스터링을 위한 입력으로 사용됩니다.
아래에 그 과정을 수학적, 구조적으로 자세히 설명드리겠습니다.
1. LSTM 입력 데이터 구성
- 유전자 발현 데이터는 다음과 같은 형태를 가집니다:
유전자\샘플 Sample1 Sample2 Sample3 ... SampleT Gene1 2.3 3.1 2.9 ... 3.2 Gene2 0.5 0.6 0.7 ... 0.8 ... ... ... ... ... GeneN 1.2 1.3 1.1 ... 1.0 - 이 데이터를 시계열로 간주하여 각 유전자(Gene_i)의 발현값 시퀀스 [x₁, x₂, ..., x_T]를 LSTM에 입력합니다.
2. LSTM 아키텍처 구성
기본 구조:
- LSTM은 순환 신경망(RNN)의 확장으로, 긴 시퀀스에서의 **장기 의존성(long-term dependency)**을 학습할 수 있습니다.
수식 (논문 기준):
LSTM은 다음과 같은 내부 연산을 통해 입력 시퀀스를 잠재 벡터로 변환합니다:
입력 게이트
$i_t = \sigma(W_i x_t + U_i h_{t-1} + b_i)$
망각 게이트
$f_t = \sigma(W_f x_t + U_f h_{t-1} + b_f)$
셀 상태 후보
$\tilde{C}_t = \tanh(W_c x_t + U_c h_{t-1} + b_c)$
셀 상태 업데이트
$C_t = f_t \odot C_{t-1} + i_t \odot \tilde{C}_t$
출력 게이트 및 hidden state
$o_t = \sigma(W_o x_t + U_o h_{t-1} + b_o)$
$h_t = o_t \odot \tanh(C_t)$
- 여기서:
- x_t: t번째 시간 샘플의 입력 (유전자 발현값)
- h_t: 해당 시점의 출력 (hidden state)
- C_t: 메모리 셀 상태
- σ \sigma: 시그모이드 함수
- ⊙ \odot: 요소별 곱
3. 유전자 임베딩 생성 방식
각 유전자 GiG_i는 길이 T인 시퀀스 $[x_1^i, x_2^i, ..., x_T^i]$를 가집니다.
이를 LSTM에 입력하면, 마지막 hidden state $h_T \in \mathbb{R}^d$가 해당 유전자의 시계열 임베딩이 됩니다.
✅ 이 임베딩은 다음을 포함합니다:
- 발현량의 변화 양상
- 패턴의 주기성, 상승/하강 추세
- 급격한 변화 등 시계열적인 특성
📌 예시 (개념적 시각화)
Gene1: [2.1, 2.3, 2.5, 2.7, 2.9] → LSTM → h_T ≈ [0.32, -0.17, 0.84, ..., 0.45]
Gene2: [0.9, 1.2, 1.4, 1.5, 1.3] → LSTM → h_T ≈ [-0.21, 0.65, -0.34, ..., 0.12]
이 벡터들이 유전자 간의 비교 및 클러스터링에 사용됩니다.
4. 반복 최적화 및 역할
- 이 LSTM 임베딩은 초기 클러스터링에 사용됨 → 이후 SVM이 클러스터를 재정비함
- 재정비된 클러스터를 바탕으로 다시 LSTM 학습 → 더 정제된 임베딩 생성
- 이 과정을 반복하여 최적의 시간 의존 기반 바이클러스터 도출
요약
| 단계 | 설명 |
| 입력 | 유전자별 시계열 발현값 |
| 모델 | LSTM |
| 출력 | 시간 패턴을 반영한 고차원 임베딩 (h_T) |
| 목적 | 클러스터링용 특징 추출 (SVM 입력) |
2. 초기 클러스터링 (Biclustering Initialization)
- LSTM 임베딩을 기반으로 유전자들을 클러스터링하여 초기 바이클러스터(bicluster) 생성
이 논문의 핵심인 Biclustering (바이클러스터링) 개념은 일반적인 클러스터링과는 다릅니다.
🔍 Biclustering이란?
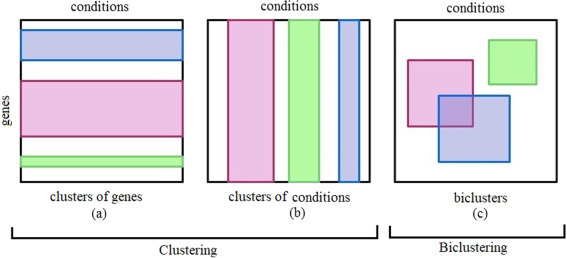
유전자(행)와 샘플(열)을 동시에 클러스터링하는 기법입니다.
즉,
- 특정 유전자들이
- 특정 샘플(환자)들에서만
- 유사한 발현 패턴을 보이는 부분 집합을 함께 묶는 것입니다.
이런 **"유전자-샘플의 이중 서브그룹"**이 바로 바이클러스터입니다.
📊 일반 클러스터링 vs. Biclustering
구분 일반 클러스터링 Biclustering| 대상 | 전체 유전자 또는 전체 샘플 | 유전자 × 샘플 서브셋 |
| 예시 | 유전자 전체를 5개 그룹으로 나눔 | 유전자 1 |
| 특징 | 한쪽(행 or 열)만 고려 | 행(유전자)과 열(샘플)을 동시에 고려 |
| 사용 목적 | 전체 유전자 또는 샘플 분류 | 특정 환자군에서만 활성화되는 유전자 집합 탐색 |
🧬 예시 (개념)
Sample1 Sample2 Sample3 Sample4 Sample5| GeneA | 5.1 | 5.3 | 5.2 | 1.0 | 0.9 |
| GeneB | 5.0 | 5.1 | 5.2 | 1.1 | 0.8 |
| GeneC | 0.5 | 0.6 | 0.7 | 0.8 | 0.9 |
- GeneA, GeneB는 Sample1~3에서만 비슷하게 높게 발현됨 → 하나의 bicluster
- 전체 유전자/샘플로 보면 이 패턴은 숨겨져 있음
📌 이 논문에서의 Biclustering
이 논문에서는 다음과 같이 biclustering을 수행합니다:
- LSTM → 유전자 임베딩 생성
- SVM으로 bicluster를 반복 최적화
- 유사한 발현 패턴을 가지는 유전자들을 클러스터링
- 동시에 이 패턴이 공통적으로 나타나는 샘플 부분집합도 함께 식별
즉, "어떤 유전자들이 어떤 환자 집단에서 함께 의미 있는 패턴을 만드는가?"를 찾는 것이 목적입니다.
🎯 왜 Biclustering이 중요한가?
- 유전자들은 모든 샘플에서 항상 함께 움직이지 않습니다.
- 특정 질병군, 세포 유형, 조건에서만 공동 발현(co-expression) 하는 경우가 많습니다.
- Biclustering은 이런 국소적 발현 패턴을 포착하기에 탁월합니다.
- 특히 암, 면역반응, 감염 질병 분석 등에서 효과적입니다.
✅ 정리
항목 내용| 정의 | 유전자-샘플의 부분 매트릭스에서 유사한 패턴을 찾는 클러스터링 |
| 역할 | 특정 유전자들이 특정 환자 집단에서만 보이는 공동 발현 패턴 탐색 |
| 이 논문에서 | LSTM 임베딩 + SVM을 이용하여 bicluster를 반복 최적화 |
| 활용 | 표현형 예측, 바이오마커 탐색, 질병 아형(subtype) 분류 등 |
3. SVM을 통한 클러스터 정제
- 각 클러스터에 대해 SVM을 적용해, 클러스터 내 유전자들의 결정 경계를 학습
- RBF 커널과 같은 비선형 커널을 사용하여 정제된 클러스터 구성
- SVM은 클러스터의 경계를 최적화하여 불일치 유전자 제거 및 재할당
논문에서 설명한 SVM을 통한 클러스터 정제 과정은 Biclustering 과정 중 유전자 클러스터의 품질을 높이기 위한 핵심 단계입니다. 여기서는 초기 클러스터링 결과를 SVM으로 다시 다듬고, 더 일관성 있는 유전자 그룹을 만들기 위해 다음과 같은 순서로 동작합니다.
🔧 1. 목적 요약
LSTM이 추출한 임베딩 기반으로 클러스터를 구성한 후,
**SVM(Support Vector Machine)**을 사용하여
**"그 클러스터가 실제로 잘 구분되는가?"**를 학습하고,
클러스터의 경계를 다듬거나 유전자를 재할당합니다.
⚙️ 2. 정제 과정 단계별 설명
🧩 초기 상태:
- LSTM → 유전자마다 시계열 임베딩 생성
- 이 임베딩으로 클러스터링 (예: K-means) → 초기 클러스터 구성됨
🧠 Step 1: SVM 모델 학습
- 각 클러스터를 이진 분류 문제로 간주
→ "이 유전자는 클러스터 A에 속하는가, 아닌가?"
예:
- 클러스터 A vs. 나머지
- 클러스터 B vs. 나머지 … 등
- 각 클러스터마다 SVM 분류기를 학습
- 입력: LSTM 임베딩 벡터
- 라벨: 클러스터 소속 여부 (1 or 0)
- 커널: RBF (Radial Basis Function) 등 비선형 경계 가능
🧠 Step 2: 유전자 재분류
- 학습된 SVM을 이용해, 각 유전자의 클러스터 소속 가능성을 다시 예측
- 예측 결과가 기존 클러스터와 불일치할 경우, 해당 유전자를 재배치하거나 제외
📌 즉:
- SVM은 **각 클러스터의 결정 경계(decision boundary)**를 학습
- 이 경계를 기준으로 클러스터 외부에 가까운 유전자는 제거하거나 재배치
🔁 Step 3: 반복 최적화 (Iterative Refinement)
- 클러스터 재조정 후, 새로운 클러스터 정보를 바탕으로:
- LSTM → 재학습 (업데이트된 클러스터 기반)
- SVM → 다시 학습하여 재정제
- 이 과정을 수렴(convergence) 조건까지 반복
🎯 예시 이미지로 표현한다면:
초기 클러스터: SVM 학습 및 정제:
[ Gene1 • ] [ Gene1 • ] ← 클러스터 A 유지
[ Gene2 • ] [ Gene2 ] ← 분류경계 밖 → 제거
[ Gene3 • ] [ Gene3 • ] ← 클러스터 A 유지
✅ 요약표
단계 설명| 1. 클러스터 초기화 | LSTM 임베딩 + 기본 클러스터링 (예: K-means) |
| 2. SVM 학습 | 각 클러스터마다 이진 분류기로 학습 (속함/안 속함) |
| 3. 유전자 평가 | 유전자의 분류 신뢰도 or 거리 기반 재배치 |
| 4. 반복 | 클러스터 → LSTM 업데이트 → 다시 SVM 반복 |
⚠️ 왜 SVM인가?
- 결정 경계를 명확하게 학습할 수 있는 강력한 분류기
- 비선형 패턴도 반영 가능 (RBF 커널 등)
- **과적합 제어 (regularization C)**로 불확실한 유전자에 대해 유연하게 대응
🔍 클러스터 정제 결과
효과 설명| 🎯 정확도 향상 | 유전자 그룹이 더 일관되고 생물학적으로 의미 있게 됨 |
| 🔎 해석 가능성 증가 | 클러스터별 특징 유전자 해석이 쉬워짐 |
| 📉 노이즈 감소 | 경계에 애매한 유전자 제거 가능 |
4. LSTM-SVM 반복 최적화 (Iterative Refinement)
- 클러스터링 결과를 바탕으로 다시 LSTM을 학습 → 새 임베딩 생성 → 다시 SVM으로 클러스터 정제
- 수렴 조건까지 반복하여 정확도와 일관성 개선
📈 성능 검증 및 결과
- 실험 환경:
- LSTM 유닛 수: 64, 입력 차원: 30
- SVM 커널: RBF, 정규화 파라미터 C=1
- 비교 대상: HMM, RNN, SVM
- 성능 지표:
- 정확도(Accuracy), 정밀도(Precision), 재현율(Recall), F1-score, 실루엣 점수
- 결과 요약:
- Accuracy 약 10% 향상
- Recall 7%, F1-score 8%, Silhouette score 9% 증가
- 계산 시간도 우수한 수준으로 유지
바이클러스터링의 생물학적 해석
- 특정 조건에서 유사한 발현 패턴을 보이는 유전자 그룹 식별
- 예: Gene1, Gene5, Gene12가 Sample2,3,7에서 유사한 패턴을 보여 바이클러스터로 분류됨
- 이들은 공통 생물학적 경로 또는 조절 메커니즘을 공유할 가능성 있음
결론 및 향후 방향
- LSTM-SVM 하이브리드는 시계열 유전자 발현 데이터에 적합한 강력한 예측 모델로 작동
- 정밀의료(Personalized Medicine)에서 치료 표적 발견, 진단 최적화에 기여 가능
- 향후:
- 실제 의료 데이터 적용
- 단일세포 시퀀싱 외에도 메타볼로믹스, 프로테오믹스 등 다양한 omics 데이터로 확장 가능
🔍 핵심 목표 요약
"유사한 발현 패턴을 보이는 유전자들(Gene)을 바이클러스터링하여 묶고,
이러한 유전자 패턴이 나타나는 환자(Sample)는 특정한 표현형(질병 등)을 가질 것이라고 예측하는 것"
📌 자세히 풀어보면:
① Gene 중심의 분석 (LSTM + SVM)
- 유전자 발현 데이터를 기반으로,
비슷한 발현 양상을 가진 유전자들을 LSTM 임베딩 → SVM으로 클러스터링 - 즉, ‘함께 움직이는 유전자 그룹(바이클러스터)’을 찾아냅니다.
📌 이 바이클러스터는 의미 있는 생물학적 단위가 될 수 있습니다.
예:
- 면역반응 관련 유전자 그룹
- 세포분열 조절 유전자 그룹
- 암세포에서만 특이적으로 활성화되는 유전자 집합
② Sample(환자) 중심의 예측 (패턴 기반 표현형 분류)
- 각 환자(샘플)의 발현 데이터를 기준으로
위에서 찾은 유전자 패턴이 얼마나 강하게 나타나는지를 측정 - 이 정보를 기반으로 환자가 어떤 표현형(질병 상태 등)에 속하는지 예측합니다.
예:
- 어떤 환자의 발현 패턴이 바이클러스터 A와 매우 유사하다면,
→ 이 환자는 A 패턴에 연관된 질병 가능성↑
정리: 두 단계로 구성된 예측 체계
| 1단계 (Unsupervised) | LSTM+SVM으로 유전자 바이클러스터 생성 (유전자 간 패턴 기반) |
| 2단계 (Supervised) | 샘플에 대해 이 바이클러스터가 얼마나 강하게 나타나는지를 계산하여 표현형 예측 |
📈 결과적으로 이 모델이 할 수 있는 일
- 의미 있는 유전자 그룹 찾기 (바이클러스터링)
→ 생물학적 경로, 조절 기작 해석 가능 - 개별 환자의 유전자 프로필로 표현형(질병) 예측
→ 정밀의료 기반 개인 맞춤 진단 가능 - 예측에 영향을 미친 유전자/클러스터 해석 가능
→ 해석 가능성 (interpretability) 보장

