Hierarchical Marker Genes Selection in scRNA-seq Analysis
Paper
https://journals.plos.org/ploscompbiol/article?id=10.1371/journal.pcbi.1012643
Hierarchical marker genes selection in scRNA-seq analysis
Author summary In the analysis and interpretation of scRNA-seq data, one important step is to identify marker genes to annotate cell clusters with the biologically meaningful names. Existing marker gene selection methods typically perform differential expr
journals.plos.org
기존 방법(one-vs-all)의 한계:
- 일반적으로 특정 cell cluster와 나머지 모든 cluster를 비교하여 marker gene을 선정.
- 그러나 서로 유사한 세포 유형(예: Naive CD4 T cell vs Memory CD4 T cell)을 분리하는 데 한계가 있음.
- 이들 사이에 공통적으로 발현되는 gene들이 marker로 선택되어, 구분력 부족과 해석 어려움을 초래함.
Methods
이 스코어링 함수는 세포 클러스터 그룹을 새롭게 정의하는 것을 목표로 하며,
이러한 세포 클러스터 그룹을 기반으로 단일 대 전체 마커 유전자가
해당 세포 클러스터 그룹에 매우 특이적이고 다른 세포 클러스터 그룹에서는 발현량이 낮도록 합니다.
어떻게 병합하나요?
- 현재 세포 클러스터들이 있다고 칩시다 (예: A, B, C, D).
- 가능한 모든 쌍 (A-B, A-C, ..., C-D)을 하나로 합쳐보고, 그 상태에서 marker gene을 선택 → 스코어 계산
- 스코어가 가장 좋아지는 병합 쌍을 선택해서 병합
- 다시 남은 클러스터로 반복
- 더 이상 스코어가 좋아지지 않으면 종료
이 과정은 사실상 **계층적 클러스터링(hierarchical clustering)**입니다. 단, 유전자 발현 패턴 기반 스코어로 병합 여부를 판단한다는 것이 핵심 차별점입니다.
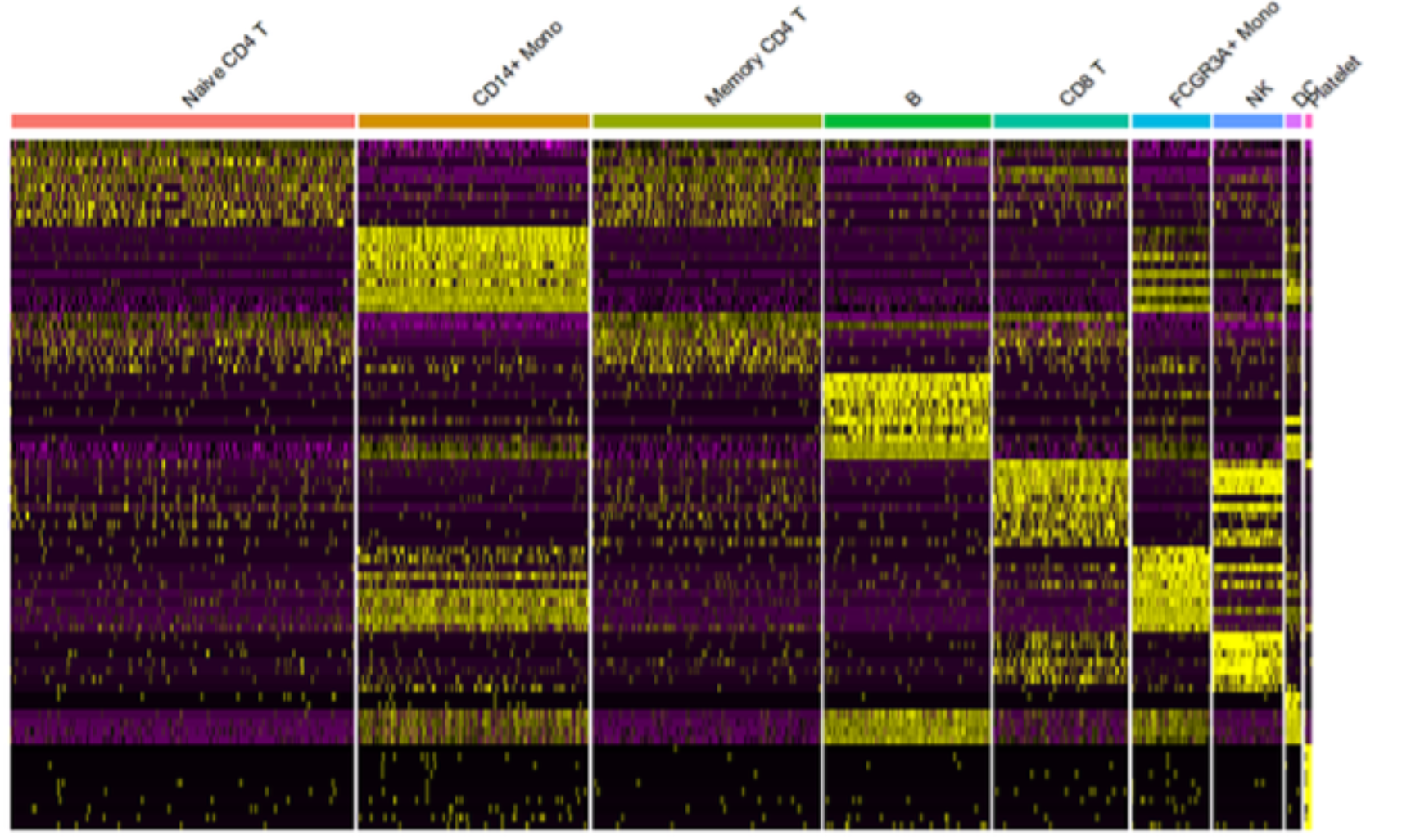
Assembled Heatmap (조립형 열지도)
이 방법의 가장 큰 장점은:
- **각 단계(split)**에서 해당 그룹을 잘 구분하는 marker gene을 찾아낸다는 점입니다.
- 각 단계마다 heatmap을 만들어 **“이 split을 잘 설명하는 유전자들”**을 보여줍니다.
어떻게 생겼나요?
- 여러 개의 split이 생기면, 그때그때 marker gene을 찾고 heatmap을 만듭니다.
- 마지막에 모든 heatmap을 위에서 아래로 차곡차곡 쌓은 것처럼 합칩니다.
- 각 가로 블록은 “특정 split에 해당하는 marker gene 열지도”입니다.
- 예: T세포 vs B세포 → 관련 유전자만 밝게 보임
- 다른 세포군에는 해당 유전자가 0 (흰색)
예시로 다시 정리
상황: Naive CD4 T와 Memory CD4 T는 서로 매우 비슷함
- 일반적인 one-vs-all 방식:
- 이 두 집단 모두에서 발현되는 유전자를 marker로 선택할 가능성 ↑
- 서로를 구분하는 데 어려움
- 계층적 방식:
- 처음에는 둘을 하나의 그룹으로 묶고 다른 세포들과 비교 → 공통 marker를 찾음
- 이후, 둘을 다시 나눔 → 이때야말로 서로를 구분하는 세밀한 marker gene을 찾게 됨
사용 데이터셋
- PBMC3k, PBMC Control, PBMC Stim
- Pancreas dataset (control case: 계층 구조가 없음)
비교 방법
- 기존 방식: All genes / Highly Variable Genes / Seurat(one-vs-all) / scGeneFit(flat & hierarchical)
- 평가 지표:
- KNN 기반 classification accuracy
- UMAP 시각화 (cell type 분리 정도)
- cross-dataset prediction accuracy (cell type mapping)
주요 결과
- 계층적 방법은 Seurat one-vs-all보다 5~15% 정확도 향상.
- UMAP 시각화에서도 더 잘 분리된 cell type cluster 확인됨.
- 서로 다른 데이터셋 간 cell type mapping 성능도 향상됨.
물학적 해석 가능성
- 계층 구조 덕분에 각 split에 대해 의미 있는 marker gene을 도출 가능.
- 예: Naive CD4 T에 대해 기존에는 포착 못했던 RPL21, Memory CD4 T에 대해 S100A11 등 세부 subtype을 구분하는 marker gene 발굴 가능.
이 논문의 궁극적인 목표는 단순히 한 가지가 아니라, 두 가지를 함께 달성하는 데 있습니다. 하지만 우선순위와 중심은 분명히 있습니다.
핵심 목표:
"더 정확하고 해석 가능한 세포 클러스터 그룹의 marker gene을 정의하는 것"
→ 즉, 세포 클러스터 단위의 marker gene 정의가 주된 목적입니다.
왜 "세포 클러스터 그룹" 정의에 집중하나요?
기존 one-vs-all 방식은:
- 각 클러스터마다 marker gene을 찾지만,
- 유사한 클러스터끼리 구별이 잘 안 됨
→ 공통된 유전자가 marker로 뽑혀버려서 해석이 어려움
그래서 이 논문에서는:
- 유사한 클러스터들을 먼저 묶고,
- 각 단계에서 적절한 marker gene을 찾아냅니다.
→ 이런 과정을 통해 "클러스터 또는 클러스터 그룹"을 설명하는 marker gene을 정의합니다.
그렇다면 "각 세포마다 marker gene"은 안 찾는 건가요?
맞습니다.
- 이 논문의 목적은 "개별 세포(cell)-단위의 marker gene"을 직접적으로 정의하는 것이 아닙니다.
- 대신, 세포들이 속한 클러스터 or 계층적 그룹 단위로 marker gene을 찾습니다.
즉,
- "이 세포가 발현한 gene이 뭐야?"가 아니라
- "이 그룹(혹은 클러스터)을 잘 구분하는 gene이 뭐야?"가 핵심입니다.
최종 정리
| 세포 클러스터 그룹을 정의하는 것이 목표인가요? | ✅ 네, 그리고 각 클러스터/그룹을 설명하는 marker gene을 정의하는 것이 주된 목표입니다. |
| 각 세포의 marker gene을 정의하는 것이 목표인가요? | ❌ 아니요. 이 논문은 개별 세포가 아닌, 세포 그룹/클러스터 수준에서 marker gene을 찾는 데 중점을 둡니다. |
즉, 이 논문은 다음을 목표로 합니다:
“유사한 세포 집단을 구분할 수 있는 계층적 marker gene을 뽑아서, 해석이 쉽고 구분이 잘 되는 세포 클러스터 구조를 만들자.”