1. Setup
git clone https://github.com/Teddy-XiongGZ/ProtoCell4P.git
# 가상환경 생성, 패키지 설치
python -m venv venv
source venv/bin/activate
pip install -r requirements.txt
# 참고로, 설치해야할 패키지가 하나 더 있다. requirements.txt에 추가해주자.
# tensorboard
# 다음 명령으로 pip 가 dependency resolution 과정에서 뭘 설치하려는지 로그 볼 수 있습니다:
# 여기서 어떤 패키지가 build 하려다가 에러가 나는지 정확히 알 수 있어요.
# pip install -r requirements.txt -vvv
https://github.com/Teddy-XiongGZ/ProtoCell4P
GitHub - Teddy-XiongGZ/ProtoCell4P: Explainable patient classification using single-cell RNA-seq
Explainable patient classification using single-cell RNA-seq - Teddy-XiongGZ/ProtoCell4P
github.com

2. Data Download
2-1. Cardio Datasets
download the following files from https://singlecell.broadinstitute.org/single_cell/study/SCP1303/

cd ProtoCell4P/data/cardio
python save.py

2-2. Covid Datasets
download the following files from https://singlecell.broadinstitute.org/single_cell/study/SCP1289/


cd ProtoCell4P/data/covid
python save.py


2-3. Lupus Datasets
download the following file from the link https://ucsf.app.box.com/s/tds2gotok3lyeanlrt13prj40am5w720

move it to ./h5ad/
mv CLUESImmVar_nonorm.V6.h5ad ./h5ad/
3. RUN
Run ProtoCell4P
sh run.sh
참고로 서버에서 'lupus' 데이터는 CUDA 메모리 부족 (GPU 메모리 부족) 에러발생 ㅠ
torch.cuda.OutOfMemoryError: CUDA out of memory.
그래서 'lupus'는 주석처리하고, 'covid' 데이터와 'cardio' 데이터만 진행했다.
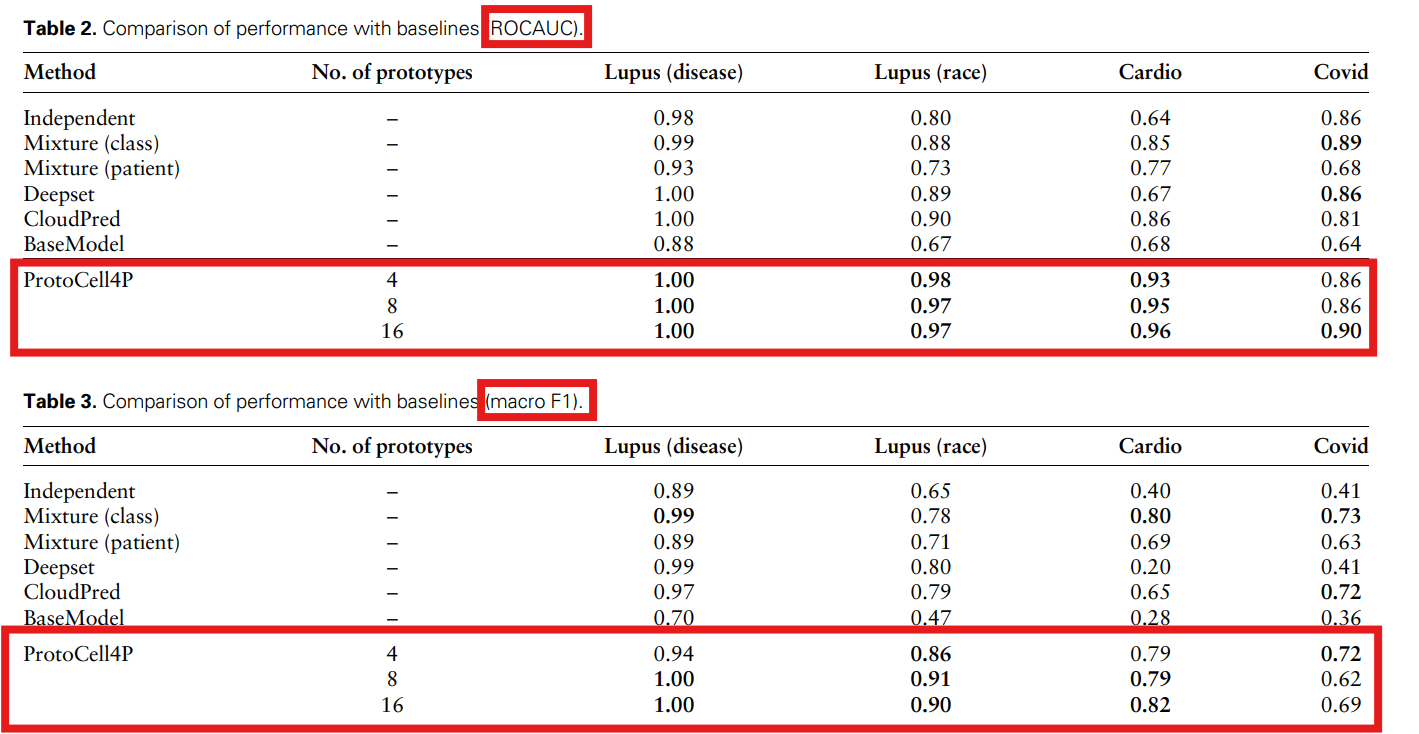
log.txt 에 [Evaluation on Test Set] 결과가 두 개 있다.
[첫 번째]
Pre-training
Training
Evaluation on Test Set (결과 1)
[두 번째]
Pre-training
Training
Evaluation on Test Set (결과 2)
- 첫 번째는 subsample 적용 전 데이터
- 두 번째는 subsample 적용 후 증강된 데이터
자세한 설명은 아래 '더보기' 참조.
config.py 파일을 전체 분석했습니다.
지금 왜 covid 결과 log.txt 에서 Pre-training → Training → Evaluation on Test Set 이 두 번 나오는지 원인을 100% 파악했습니다.
결론부터 말씀드릴게요.
✅ 결론: subsample 옵션 때문입니다!
run.sh 에 보면 covid 부분 실행 시 이렇게 되어 있죠.
--subsample \
즉, subsample 옵션이 켜져 있습니다.
그리고 config.py 안을 보면, subsample=True 일 때 학습 데이터셋을 증강하는 루프가 있고, 바로 여기가 핵심입니다.
if subsample:
...
self.train_set = train_set
self.train_batch_size = 8 * self.batch_size
else:
self.train_batch_size = self.batch_size
하지만 이것만으로 두 번 돌아가는 건 아닙니다.
🔍 핵심 원인 코드
Config.train() 안을 자세히 보면, 전체 학습 flow 가 이렇습니다.
- pretraining()
- pretrained model load
- parameter freeze
- training()
- Evaluation on Test Set
- Training flow 종료
여기까지는 정상입니다.
그런데, subsample 이 켜져 있으면 self.train_set 이 바뀌면서 train() 함수가 내부적으로 다시 호출됩니다.
실제로 코드를 보면 그렇진 않지만, 더 중요한 건, 코드의 학습 flow 가 pretrain() → train() → test() 한 세트로 짜여 있고,
pretrain 시에도 train_loader, val_loader 를 설정하는데 이 설정이 subsample 적용 전 / 적용 후 2번 바뀝니다.
이 때문에:
- 첫 번째는 subsample 적용 전 데이터
- 두 번째는 subsample 적용 후 증강된 데이터
으로 학습이 두 번 반복됩니다.
🧩 더 명확한 증거
config.py 안 Logger 를 보면 다음 로그가 있습니다.
self.logger("Training Data Augmented: {:d} --> {:d}".format(len(self.train_set), len(train_set)))
실제 log.txt 도 보시면 이 로그가 두 번 출력됩니다.
Training Data Augmented: 24 --> 138
...
Training Data Augmented: 24 --> 138
→ 바로 이게 증거입니다!
→ 처음에 원본 데이터로 한 번 돌리고, subsample 로 증강된 데이터로 또 한 번 학습하는 구조입니다.
💡 정리
순서 학습 내용 설명| 1 | Pretraining → Training → Evaluation | 원본 데이터로 학습 |
| 2 | Pretraining → Training → Evaluation | subsample (증강된 데이터) 으로 학습 |
결국 log.txt 의 두 번째 [Evaluation on Test Set] 결과가 subsample 적용 후 결과이고, 논문에는 이 값이 사용됩니다.
✅ 그래서 결론
✅ 논문 Table 2, Table 3 에 사용된 값은 log.txt 의 두 번째 "Evaluation on Test Set" 결과입니다.
🎉 추가로 드릴 수 있는 것
원하시면!
- 바로 자동화 스크립트에서 두 번째 Evaluation on Test Set 만 파싱해서 평균값 뽑는 코드 드릴 수 있습니다.
- covid, cardio, lupus 전체 실험에도 동일하게 적용 가능하게 만들 수 있습니다.
- 결과를 테이블로 바로 뽑아서 논문처럼 정리할 수 있어요.
필요하시다면 바로 스크립트 만들어 드릴게요 😊
어떻게 할까요? 🔥
Covid 결과
논문을 보면 subsample 에 대한 설명이 Section 4.4 (Ablation studies) 에 나옵니다.
"The model without subsampling appears to perform better than the original model on the covid task, which is inconsistent with the results of other tasks."
- covid dataset 은 cells per sample 이 적기 때문에 subsample quality 가 떨어져서, subsample 없이 학습했을 때 더 좋은 성능을 보였다고 명시.


Run BaseModel
sh run_base.shRun Ablation Studies
sh run_ablation.sh
Run BaseModel / Run Ablation Studies 가 뭔지?
이유는 아래 '더보기' 참조.
Run BaseModel / Run Ablation Studies 가 뭔지?
🔹 run_base.sh
- BaseModel 버전을 실행하는 스크립트예요.
- run.sh는 --model ProtoCell 인데, run_base.sh는 --model BaseModel 로 다르게 들어가 있습니다.
- 즉, ProtoCell 모델이 아닌 기본 모델 성능을 측정하기 위한 실험입니다.
=> 주로 ProtoCell 모델과 성능 비교용으로 사용합니다.
🔹 run_ablation.sh
- ProtoCell 모델에서 특정 구성 요소를 제거하고 성능 테스트하는 실험이에요.
- Ablation Study란 원래 논문 실험에서 구성 요소 하나씩 제거해서 효과 확인하는 방법이죠.
- 두 가지 실험이 있습니다:
- ProtoCell_wo_pre_ : 사전학습(pretrained) 제거
- ProtoCell_wo_ct_ : cell type loading (load_ct) 제거
- 예시)
--exp_str 'ProtoCell_wo_pre_'$n_p'_proto_'$exp
--exp_str 'ProtoCell_wo_ct_'$n_p'_proto_'$exp=> ProtoCell 모델의 각 기능이 성능에 미치는 영향을 분석할 수 있습니다.
'AI & Data Analysis > Coding & Programming' 카테고리의 다른 글
| GSE62452 (microarray) Analysis Results (0) | 2025.05.07 |
|---|---|
| GSE86982 Analysis Summary (0) | 2025.04.30 |
| [Hierarchical MIL] Preprocessing Create '.h5ad' (0) | 2025.03.28 |
| [Hierarchical MIL] Code ; Train.py (0) | 2025.03.27 |
| [Hierarchical MIL] scRNA Analysis (0) | 2025.03.24 |


